A vida enquanto um processo químico auto-organizado de codificação
Introdução
Ao longo da última década, estivemos trabalhando com um dos grandes mistérios da humanidade: a origem da vida. É incrível constatar como fizemos grandes avanços teóricos nesse campo a partir do questionamento de certos conceitos tidos como fundamentais na área.
A questão sobre “o que é a vida?” é provavelmente umas das mais importantes do campo de pesquisa conhecido como filosofia da biologia. Ao lado dela, outras questões importantes também se colocam, tais como: “o que é um gene?”, “o que é um indivíduo?”, “o que é uma espécie?”, dentre outras. Mas é possível dizer que “o que é a vida?” seja mesmo a questão mais relevante que uma pessoa pode se fazer quando se põe a pensar sobre os organismos vivos.
Diz-se que há tantos conceitos de vida quanto pessoas que se colocam esta pergunta e, de fato, o conceito de vida está muitas vezes relacionado a uma cosmovisão mais ampla compartilhada por indivíduos e grupos sociais, inclusive com vieses religiosos e políticos. Entretanto, mesmo entre especialistas e cientistas que trabalham há décadas na área, há ainda muita confusão entre os conceitos e muitas controvérsias. Fato é que, dentro do paradigma contemporâneo da biologia, tem-se considerado que a unidade básica da vida é uma célula. Há muitas gerações os biólogos são educados dessa forma, mas há questões importantes que desafiam tal concepção.
Além da abordagem clássica que considera (i) a vida enquanto celular, outras abordagens consideram (ii) a vida enquanto moléculas autorreplicantes ou enquanto um (iii) sistema complexo operando no limite do caos. Analisamos todas essas teorias há algum tempo (Farias, Prosdocimi e Caponi, 2021) e refutamos todas elas. Resumindo brevemente nossa argumentação, se está claro que a vida é mesmo um sistema complexo, existem muitos sistemas complexos não vivos, tais como furacões, tempestades solares, explosões de supernovas e outros. Por outro lado, embora a vida seja formada por moléculas autorreplicantes, a autorreplicação per se deve ser considerada como um tipo de ciclo bioquímico e pode ser encontrada em sistemas cíclicos não-vivos que repetem padrões ordenados. Finalmente, existem sistemas biológicos complexos e capazes de evolução que são mais simples que as células, tais como os vírus em geral e, em particular, os vírus gigantes.
Para criarmos um bom conceito que defina categoricamente “o que é a vida?” precisamos que ele seja necessário e suficiente. Do contrário, criamos um conceito tão amplo que pode permitir enquadrar diferentes entidades dentro dele, não se tornando verdadeiramente útil. Por outro lado, se fechamos demasiadamente um conceito, ele deixa de representar a verdadeira diversidade das entidades que almejamos conceituar.
A vida sem dúvida é um sistema complexo e é baseada em moléculas autorreplicantes, mas esses conceitos não são suficientes para definir a vida (Prosdocimi, Jheeta e Farias, 2018). Por outro lado, a célula é sem dúvida um sistema vivo, mas não nos parece necessário termos um sistema já celularizado para definirmos a vida tal qual conhecemos aqui no planeta Terra. Como a vida terráquea é a única que conhecemos, a maioria das questões que se coloca em termos do campo da astrobiologia consiste em especulações ainda bastante controversas.
Uma questão que coloca quando analisamos a vida na Terra é que os sistemas biológicos mais abundantes em nosso planeta são os vírus. E, curiosamente, a teoria paradigmática da biologia os define como não vivos ou, algumas vezes, considera que estejam vivos quando estão dentro das células se reproduzindo e “sequestrando” a maquinaria celular em seus “propósitos egoístas”. Ora, os cálculos mais recentes consideram que a entidade biológica mais abundante na Terra são os vírus. Sugere-se que há mais vírus no planeta Terra do que estrelas em toda o universo conhecido! O número estimado de vírus é tão alto quando 1023 (Suttle, 2013). Mesmo que este número esteja superestimado, a quantidade de vírus existentes no planeta é incrivelmente maior do que a quantidade de células (Bergh et al., 1989). Com tanta abundância, é de se imaginar que uma grande parte do que os biólogos têm chamado de virosfera seja desconhecida (Comeau et al., 2008). Os pesquisadores têm muitas vezes se referido a esse grande volume como a questão da matéria escura viral (Roux et al., 2015). Além disso, a relevância dos vírus para os ecossistemas foi negligenciada por muito tempo (Prosdocimi e Farias, 2019; Prosdocimi e Farias, 2020; Prosdocimi et al., 2023). Hoje, porém, sabemos que eles são incrivelmente abundantes principalmente em sistemas aquáticos e, em particular, nos oceanos. Ecologicamente falando, reconhece-se hoje que os vírus funcionem no controle de populações de bactérias e outros hospedeiros (Koskella et al., 2022), na ciclagem de nutrientes, principalmente do carbono (Weitz and Wilhelm, 2012), e no embaralhamento de genes dentro de genomas de organismos mais complexos (Enard et al., 2016). Outra de nossas bases de apoio ao longo da presente linha de raciocínio é que os vírus não podem ficar de fora quando almejamos definir “o que é a vida?”
A vida como um processo
Ao começarmos a refletir sobre o assunto e verificar as principais teorias usadas para definir vida, acreditamos termos encontrado uma deficiência fundamental na própria classificação ontológica na qual os pesquisadores têm classicamente tentado enquadrar a vida. A vida tem sido entendida como formada por entidades materiais que precisam ser descritas por características específicas ou tipos naturais. Nossa proposta, entretanto, almeja considerar que a vida está baseada em um processo que ocorre através de moléculas materiais, mas que está além delas. A vida é um sistema emergente e é mais do que a soma das interações de seus componentes, de forma que, para os sistemas vivos, o todo é maior do que a soma de suas partes. Ao aplicarmos à origem da vida ideias usadas por pesquisadores da chamada teoria do caos, podemos observar que sistemas dinâmicos altamente complexos tais como os sistemas vivos podem mostrar comportamentos emergentes que não podem ser explicados apenas pela análise das partes isoladas do sistema.
Tradicionalmente, a biologia concentra seus estudos na compreensão de características tipológicas observadas nas entidades biológicas, como o estudo e classificação das diferentes espécies ou de diferentes tipos celulares. Tal método histórico dá a entender que tais características precederam os organismos e os processos que as mantêm (Dupré e Nicholson, 2018).
Muitos autores, entretanto, preferem sair da esfera das definições tipológicas para focar nos processos, uma vez que o mundo vivo apresenta uma hierarquia identificável de tais processos (Dupré, 2012; Simons 2018). Dupré e Nicholson (2018) argumentam que as entidades e suas características podem ser mais bem compreendidas enquanto estágios temporais de processos estáveis. Simons (2018) sugeriu que as entidades materiais devem ser entendidas como um tipo de precipitação dos processos que as mantêm e as estabilizam. Seguindo uma linha de raciocínio semelhante, propomos que os seres vivos devem ser compreendidos como a materialização dos processos que são essenciais para sua manutenção e estabilidade dinâmica (Farias e Prosdocimi, 2022). Mas quais seriam esses processos?
O reposicionamento do fenômeno da vida da categoria ontológica de Matéria para a categoria de Processos é essencial para nos guiar a um conceito mais adequado para a vida. Uma consequência direta dessa substituição ontológica é a modificação na maneira pela qual os seres vivos passam a ser analisados e compreendidos. Assim, precisamos buscar as leis gerais que determinem a lógica de funcionamento mais profunda dos seres vivos. Com essa concepção em mente, ao analisarmos os organismos vivos que conhecemos em nosso planeta, observamos que todos eles processam códigos em múltiplas camadas.
Biosemiótica X Biologia dos Códigos
A ideia de que a biologia é baseada em códigos é interessante e precisa ser avaliada com profundidade. Em 1998, o biólogo teórico dinamarquês Claus Emmeche propôs que o conceito de vida deveria ser baseado na natureza código-estrutural dos sistemas biológicos (Emmeche, 1998). Emmeche foi pioneiro no campo que veio a ser conhecido como biosemiótica e, inspirados por seus trabalhos, seguimos uma linha de raciocínio semelhante. Por outro lado, nossa proposta diverge das ideias de Emmeche porque sua teoria está mais relacionada à forma como os organismos biológicos se comunicam e trocam informações. Os pesquisadores em biosemiótica concentram-se em compreender como a comunicação ocorre em organismos biológicos por meio de seus sistemas codificados.
Em sua obra intitulada “Da Biosemiótica para Biologia dos Códigos” (Barbieri, 2014), o pesquisador italiano Marcello Barbieri conta a história da biosemiótica no início dos anos 2000, culminando com sua decisão de romper com o movimento. A principal questão em disputa estava relacionada ao fato de que os biosemioticistas baseavam suas ideias na interação de codificação e decodificação, na qual a interpretação dos códigos deveria ser compreendida como a característica mais importante na biologia. Por outro lado, a proposta de Barbieri estava focada na ideia de que os seres orgânicos eram produzidos por processos de codificação que operavam em nível molecular. Embora a diferença possa parecer sutil, o impacto conceitual da disputa é de extrema importância até hoje. Mudar o foco da interação de códigos de interação para a compreensão da própria essência molecular dos processos de codificação tornou a Biologia dos Códigos de Barbieri mais adequada à prática científica. Isso aconteceu porque as questões mais importantes para ele deveriam ser respondidas estudando a natureza mecanicista dos processos de codificação e decodificação observados nos seres vivos. Para entender o que é a vida, portanto, precisaríamos entender como os processos moleculares tinham se auto-organizado de fato, através de um olhar sobre as moléculas básicas que constituem a vida. É um processo filosófico, mas é baseado na observação empírica sobre como os códigos orgânicos de fato se organizam nos sistemas biológicos, ou seja, é baseado na compreensão do processo físico e químico que constitui a vida em seu nível molecular mais básico.
Quando observamos os sistemas biológicos, percebemos que várias estruturas operam em formas codificadas. Em nível molecular, poderíamos descrever, por exemplo: o código genético, a regulação da expressão gênica, o sistema de sinalização hormônio-receptor, o código das histonas, o código epigenético, e muitos outros. Em níveis mais elevados, outros códigos podem ser descritos, tais como: a linguagem, a seleção sexual, a conduta social; entre muitos outros. É importante observar que todos os sistemas de codificação que conhecemos existem apenas em sistemas biológicos ou em sistemas diretamente derivados de organismos biológicos, como no caso dos idiomas ou dos programas de computador (Farias, Prosdocimi e Caponi, 2021). Ao analisarmos a origem e a evolução dos códigos, é possível vislumbrar que a sobreposição de novos códigos sobre os antigos muitas vezes aumenta a complexidade dos códigos anteriores, operando geralmente para ajustá-los de forma mais fina e precisa. Esses códigos interrelacionados começam a interagir para formar macrocódigos compostos por múltiplos sistemas de codificação sobrepostos (Farias, Prosdocimi e Caponi, 2021; Prosdocimi e Farias, 2021). A presença e a relação integrada entre essas múltiplas camadas de codificação são características fundamentais que definem a organização dos seres vivos.
Barbieri argumentou ainda que a questão mais importante em um código estava relacionada ao fato de que as regras de codificação não deveriam ser ditadas exclusivamente pelas leis da física e da química (Barbieri, 2014). Para que um código biológico fosse estabelecido, era necessário a existência de regras arbitrárias que teriam sido auto-organizadas para permitir a correspondência entre dois mundos linguístico-moleculares independentes (Barbieri, 2003). No caso da vida que temos na terra, os mundos linguísticos mais básicos no qual a vida se baseia são: (i) o mundo dos ácidos nucleicos (DNA e RNA) e (ii) o mundo dos peptídeos ou proteínas (Prosdocimi e Farias, 2019).
Assim, na biologia, os códigos orgânicos teriam evoluído por princípios emergentes de auto-organização, permitindo a ligação codificada entre esses dois mundos moleculares de ácidos nucleicos e peptídeos previamente desconectados na Terra primitiva. Essa ligação acontece ainda hoje, dentro das células, através do mecanismo molecular conhecidos como “aparato de síntese proteica” ou “mecanismo de tradução”. Nossa proposta atual é baseada na ideia de Barbieri de que a organicidade foi construída sobre processos básicos de codificação e que tais processos devem ser descritos e compreendidos através da construção de modelos mecanicistas (Barbieri, 2014) baseados no estudo da origem deste primeiro mecanismo de tradução molecular.
Ácidos nucleicos e peptídeos/proteínas: polímeros biológicos essenciais
Como nossa teoria é baseada no funcionamento molecular mais básico dos sistemas vivos, ou seja, no funcionamento molecular do código genético, seria impossível continuarmos nossa argumentação sem apresentarmos breves informações sobre como a biologia funciona em seu nível mais molecular.
Desde que Watson e Crick descobriram que o DNA é um enorme polímero formado por uma molécula de fita-dupla antiparalela, contendo um backup de si mesma, e capaz de se replicar ao abrir-se (Watson e Crick, 1953), a biologia molecular avançou enormemente. Mais do que isso, a descoberta de Watson e Crick deixava claro que os genes seriam formados pela sequência de letras (ou nucleotídeos) do DNA. O próprio Watson reconheceu que a ideia de que o gene seria uma molécula teria sido inspirada pela obra clássica do prêmio Nobel austríaco Erwin Schrödinger (1887-1961) publicada em 1944 e intitulada “O que é a vida?” (Schrödinger, 1944; Watson, 1968). Assim, a natureza material do gene havia sido descoberta: ele tinha um conteúdo informacional definido pela sequência de nucleotídeos, da mesma forma que as palavras dependem da ordem das letras para serem entendidas. CATA é diferente de TACA, assim como é diferente de ACTA ou de uma palavra com outras letras, tal como GATA. Descobriu-se assim que os genes tinham essas sequências semânticas específicas, formadas por milhares ou milhões dos nucleotídeos de adenina, citosina, guanina e timina concatenados. E justamente a ordem das letras em uma palavra de DNA definiria o código do gene. Tal código de nucleotídeos dos genes será importante para definir um outro código, que será a sequência de aminoácidos nas proteínas (pelo menos para os genes codificadores de proteínas).
Os ácidos nucleicos são, portanto, formados por polímeros de nucleotídeos ligados como num colar de contas. Uma molécula de DNA pode ter dezenas de milhões dessas contas, como o cromossomo 1 humano, que tem aproximadamente 248.000.000 de nucleotídeos, ou ainda mais. Por outro lado, um pequeno genoma viral pode ter apenas alguns milhares de nucleotídeos. Por exemplo, o genoma do vírus Sars-Cov-2, responsável pela COVID-19, tem meros 30.000 nucleotídeos e é formado por uma molécula de RNA de fita simples. E os nucleotídeos são moléculas um tanto quanto complexas, contendo um grupo fosfato, um açúcar conhecido como ribose e uma base nitrogenada. Com aproximadamente 39 átomos, o nucleotídeo é uma molécula não tão complexa, sendo que a soma dos números moleculares de seus átomos é de aproximadamente 61 (para o nucleotídeo de adenina). Esses polímeros de nucleotídeos concatenados formam os dois principais tipos de ácidos nucleicos que conhecemos: o DNA e o RNA. Hoje sabemos que o DNA pode ser visto como uma macromolécula de armazenamento de informação mais otimizada, enquanto o RNA, que é mais simples e instável, muito provavelmente surgiu antes (Prosdocimi e Farias, 2019). De qualquer forma, DNA e RNA são as principais moléculas responsáveis pela transmissão da informação genética entre gerações, mesmo quando consideramos a importância da herança citoplasmática ou epigenética.
Já os peptídeos são compostos por polímeros de aminoácidos. Um peptídeo normalmente tem entre 1 e 100 aminoácidos encadeados. Em geral, quando um peptídeo tem mais de 100 aminoácidos, ele é chamado de proteína. E as proteínas em geral possuem centenas de aminoácidos ou, as maiores, alguns milhares de aminoácidos (a maior proteína humana é a Titina, ela pode ter até 40.000 aminoácidos, mas é uma exceção). As proteínas em geral têm, em média, 300 a 500 aminoácidos. Repare que são muito menores do que os milhões de nucleotídeos que uma molécula de DNA cromossomal pode conter.
Uma questão importante entre ácidos nucleicos e proteínas é que os ácidos nucleicos são quimicamente “insípidos”, ou seja, todas as quatro bases nitrogenadas (A, C, G e T ou U) têm características químicas parecidas. Isso é importante justamente para que o DNA não seja tão reativo e possa funcionar bem ao armazenar a informação genética. Por outro lado, as proteínas são formadas por 20 tipos diferentes de aminoácidos que são bastante diferentes com relação às suas características químicas. Além disso, tal como nos ácidos nucléicos, também nas proteínas a ordem dos tijolos moleculares ou monômeros (no caso, os aminoácidos) é importante; essa ordem forma o que chamamos de sequência primária da proteína. As proteínas, entretanto, funcionam de acordo com sua estrutura tridimensional que é produzida quando esses aminoácidos se enrolam entre si no processo de enovelamento que dá, à cada proteína, uma forma 3D particular. É essa forma que vai permitir que ela se ligue nos mais diferentes compostos. É por isso que as proteínas (e não outras moléculas) são as principais moléculas efetoras da célula, funcionando como “os trabalhadores” da cidade-célula e sendo responsáveis por organizar o metabolismo ao se ligarem a diferentes moléculas e catalisarem reações químicas.
A catálise enzimática é um fenômeno fundamental que ocorre em processos biológicos, possibilitando e acelerando reações químicas específicas. As enzimas, proteínas biológicas especializadas, atuam como catalisadores ao facilitar a conversão de substratos em produtos, sem se consumirem no processo. O processo de catálise enzimática inicia-se com a ligação de uma molécula química conhecida como substrato à enzima, formando o complexo enzima-substrato. Essa interação cria um ambiente propício para a reação química, reduzindo a energia de ativação necessária para a ocorrência do processo. A enzima desempenha um papel essencial ao orientar e estabilizar as moléculas do substrato, promovendo a colisão entre elas. Após a reação, os produtos são liberados, e a enzima, inalterada, fica disponível para participar de novas reações. Esse ciclo de (i) ligação, (ii) facilitação da reação e (iii) liberação do produto é crucial para a maioria dos processos metabólicos e reações celulares. A especificidade das enzimas para seus substratos e sua capacidade de regular vias metabólicas desempenham papéis vitais na manutenção do equilíbrio celular e na eficiência dos sistemas biológicos.
As enzimas mais importantes para o metabolismo celular são proteínas já que diversas propriedades exclusivas das proteínas favorecem a eficácia da catálise biológica. As proteínas, constituídas por sequências específicas de aminoácidos, possuem uma capacidade notável de se enovelar em estruturas tridimensionais complexas. Essa complexidade estrutural é crucial para conferir às enzimas uma alta especificidade em relação aos substratos, permitindo o reconhecimento preciso de moléculas específicas. A adaptabilidade estrutural é outra característica intrínseca das proteínas. A capacidade de modificar suas estruturas em resposta a variações ambientais é essencial para uma catálise eficaz, uma vez que possibilita que as enzimas se ajustem aos formatos dos substratos, promovendo interações mais eficientes que dependem também do meio ambiente químico em seu entorno. Além disso, sendo formadas por 20 tipos de aminoácidos diferentes, com estrutura química bastante variada, as proteínas apresentam grupos químicos de funcionalidades específicas em sua estrutura, participando diretamente nas reações catalisadas. Esses grupamentos moleculares quimicamente ativos desempenham um papel crucial na transferência de elétrons ou prótons durante as reações, executando a atividade catalítica das enzimas. Nas células, a escolha preponderante de utilizar proteínas como constituintes fundamentais das enzimas está intrinsecamente ligada às propriedades únicas dessas macromoléculas. Essa escolha definida ao longo do processo evolutivo, reflete a adaptabilidade e eficiência das proteínas na execução dos papéis catalíticos essenciais nas reações bioquímicas que sustentam a vida.
Apesar disso, existem exceções importantes à esta regra, já que algumas enzimas conhecidas como ribozimas são formadas por moléculas de RNA. Tais ribozimas também se enovelam em uma estrutura tridimensional apropriada e são capazes de apresentar atividade catalítica. Porém a diversidade química dos nucleotídeos é muito menor do que a diversidade dos aminoácidos, restringindo assim a ação das ribozimas. No início da vida, entretanto, vamos ver que as ribozimas e, em particular, uma ribozima em especial tiveram um papel fundamental para que a vida pudesse emergir.
Antes de terminar essa seção, vale a pena esclarecer o conceito de ribonucleoproteína, já que consideramos que a vida é baseada em um código ribonucleoproteico. Basicamente uma ribonucleoproteína é um aglomerado multimolecular composto de pelo menos uma molécula de ácido nucleico (seja RNA ou DNA) e peptídeos ou proteínas associadas. Nas células atuais, o DNA está sempre envolvo em moléculas proteicas, principalmente as proteínas histonas, que formam a estrutura conhecida como cromatina, que é a base para formar os cromossomos. Também a estrutura básica de um vírus consiste em um aglomerado ribonucleoproteico, onde um genoma de RNA ou DNA é envolto por proteínas do capsídeo, que formam o envelope viral. Onde quer que haja vida como conhecemos na Terra, haverão ribonucleoproteínas.
A simbiose química
Sabemos que a vida se iniciou em um ambiente pré-biótico rico de moléculas orgânicas dispersas na chamada “sopa primordial”. Esse conceito de sopa primordial foi criado na década de 1920 pelo bioquímico russo Alexandr Oparin (1894-1980) e sugere que, nas condições da Terra primitiva, compostos orgânicos essenciais para a vida poderiam ter se formado em uma “sopa” de substâncias químicas simples, impulsionadas pelas condições geoquímicas da época (Oparin, 1924). A vida emergiu quando determinadas moléculas dessa sopa iniciaram um processo de auto-organização e hoje temos uma ideia bastante sofisticada sobre quais foram essas moléculas e como isso aconteceu. Essa auto-organização avançou na medida em que moléculas de ácidos nucleicos e proteínas interagiam e se tornavam mais estáveis e mais capazes de se autorreplicarem (Prosdocimi, José e Farias, 2021). As moléculas que passaram por esse processo se mostraram, portanto, mais perenes e capazes de aumentarem sua quantidade na sopa primordial. A vida, portanto, irá emergir a partir do que chamamos de interação simbiótica entre essas duas macromoléculas fundamentais: os ácidos nucleicos e as proteínas (Figura 2). Ao longo desse processo de simbiose, ambas as moléculas participantes da interação foram beneficiadas e passam a viver juntas (Farias e Prosdocimi, 2022). Esse também é o princípio que Lanier e colaboradores chamaram de mutualismo molecular (Lanier, Petrov e Williams, 2017).
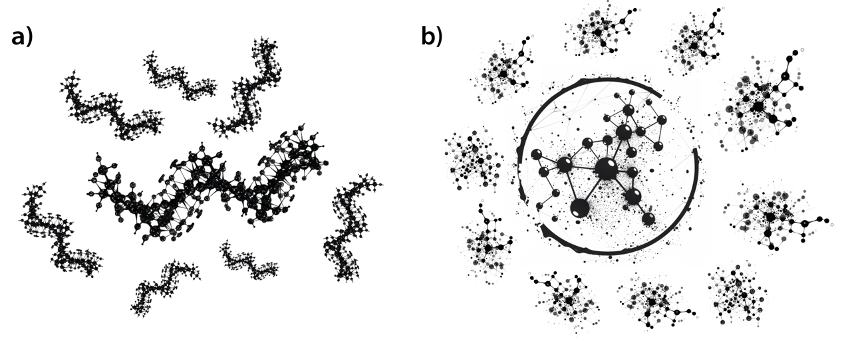
Esse processo de auto-organização e simbiose irá produzir, de uma forma que ainda não entendemos muito bem, um mecanismo molecular codificado que será capaz de “ler” a informação de um ácido nucleico e “imprimir” uma proteína correspondente a essa informação. O ácido nucleico que é de fato lido nesse processo não é o DNA, mas sim seu primo mais antigo, o RNA, que além de se apresentar mais comumente em fita simples, é formado por um açúcar do tipo ribose e um nucleotídeo de uracila (U) no lugar da timina (T). Esse processo que transforma a informação química em formato de nucleotídeos encadeados em uma informação química formada por aminoácidos encadeados foi um processo altamente complexo. Através desse processo, o código genético emergiu juntamente com o mecanismo de síntese de proteínas (ou mecanismo de tradução). Ao longo desse processo de tradução molecular automontada, cada 3 nucleotídeos (também chamados de trincas ou códons) vão codificar um, dentre os 20 diferentes tipos de aminoácidos existentes nos organismos vivos. Como existem 4 nucleotídeos, sua organização 3 a 3 produz um código com 43, ou 64 combinações possíveis. Um códon foi considerado como uma sequência específica de três nucleotídeos (uma trinca) em uma molécula de ácido nucleico (como DNA ou RNA). Códons desempenham um papel fundamental na síntese de proteínas durante o processo de tradução genética. Cada códon corresponde a um aminoácido específico ou serve como um sinal para iniciar ou terminar a tradução. Ao longo de uma década, entre 1960 e 1970, todos os 64 códons foram associados a um aminoácido específico, sendo que se descobriu que a trinca AUG, por exemplo, formava o chamado “códon de iniciação” e codificava o aminoácido conhecido como metionina, sendo usada quase sempre como aminoácido inicial de uma proteína ou peptídeo. Além disso, três trincas ou códons (UAA, UAG e UGA) não codificavam aminoácido algum e eram usadas para indicar o término da síntese de proteínas.
Nos organismos vivos hoje em dia, esse processo de tradução ou síntese de proteínas envolve três tipos de RNAs (o mensageiro, o transportador e o ribossômico) e dezenas de proteínas diferentes, de forma que é muito difícil entender como ele se auto-organizou a partir de moléculas muito mais simples presentes na sopa primordial. De alguma forma, entretanto, isso precisa ter acontecido para que estejamos hoje tentando contar esta história.
Assim, foi a emergência de um sistema de tradução molecular, que lê um ácido nucleico (códon a códon) e produz uma proteína (aminoácido por aminoácido), que permitiu o surgimento da informação genética e dos genes. Por isso, quando buscamos qual teria sido o primeiro gene, precisamos considerar que o surgimento do primeiro gene estava intrinsecamente ligado ao próprio mecanismo que cria a codificação genética (Prosdocimi e Farias, 2023). E foi esse provavelmente o primeiro código químico que permitiu à vida emergir (Prosdocimi, José e Farias, 2021) e que, posteriormente, permitiu a sobreposição de outros códigos químicos para produzir a vida como uma estrutura de macrocódigos, tal como a conhecemos hoje (Farias, Prosdocimi e Caponi, 2021).
Assim, a correspondência entre ácidos nucleicos e peptídeos ou proteínas precisou ser implementada por um tipo de tradutor ou adaptador, uma molécula capaz de efetivamente realizar a codificação molecular. Já está claro que o mecanismo de tradução deve ser entendido como sendo esse adaptador ou conversor (Wächtershäuser, 1998; Goodwin et al., 2012). Sabemos que a principal “máquina molecular” onde essa tradução acontece é uma organela chamada de ribossomo. Ela é a responsável por realizar uma conversão digital para analógica, lendo informações digitais escritas na linguagem de ácidos nucleicos (organizada em códons) e traduzindo-as em aminoácidos concatenados nas moléculas de proteínas. Tais proteínas têm informações estruturais analógicas, representadas por suas estruturas tridimensionais e superfícies eletroquímicas, capazes de ligar moléculas e catalisar reações enzimáticas. Mas não é só o ribossomo que é responsável por essa tradução, antes dele atuar é preciso que exista um outro mecanismo complexo capaz de carregar aminoácidos específicos em moléculas chamadas de RNAs transportadores. Essa seria uma preparação necessária para o mecanismo de tradução e ele é direcionado através de enzimas bastante específicas e conhecidas como aminoacil tRNA transferases (Schimmel et al., 1993). Esse processo é bastante complexo e está intimamente associado à origem do código, porém ainda não sabemos exatamente como ele poderia ter sido auto-organizado em nível molecular, na Terra primitiva.
O LUCA é o ancestral das células
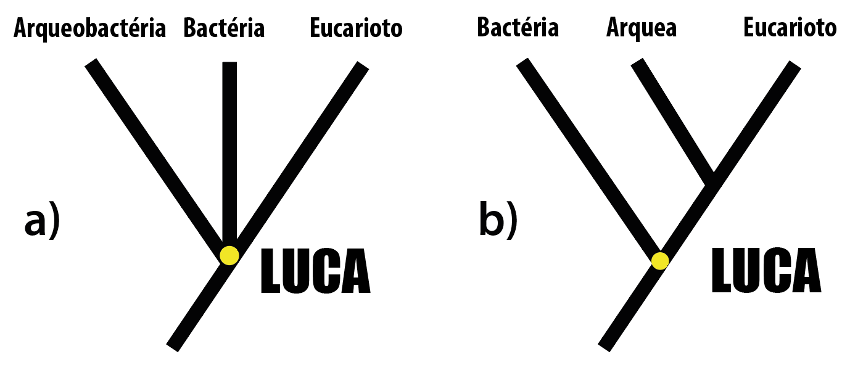
A primeira questão que nos incomodava nos modelos clássicos sobre a origem da vida era o conceito do último ancestral comum universal, também chamado de LUCA (do inglês, Last Universal Common Ancestor). O conceito de LUCA havia sido proposto pelos microbiologistas estadunidenses Carl Woese e George Fox na década de 70 (Woese e Fox, 1977a). Woese tinha feito uma descoberta fundamental à época: ele percebera que o grupo de organismos que antigamente chamávamos de bactérias (pertencentes ao reino Monera) podia ser dividido em dois grandes grupos de organismos que apresentavam características moleculares fundamentalmente divergentes. À época, o sequenciamento de DNA pelo método de Sanger ficava cada vez mais simples de ser realizado e Woese encontrou então alguns organismos procarióticos (que não possuem um envelope nuclear para guardar o material genético) que viviam em ambientes extremos e que tinham uma sequência de DNA significativamente diferente de bactérias conhecidas comumente (Woese e Fox, 1977b).
Devido ao fato de que esses organismos viviam em ambientes muito quentes ou com muita quantidade de sal, Woese pensou que eles seriam os ancestrais das bactérias comuns e, por isso, lhes batizou como arqueobactérias (archaea, do grego para “antigo”). Em 1990, Woese e Fox decidem revisitar a divisão de todo o conjunto de organismos existentes na Terra (os famosos 5 reinos) e definem uma hierarquia nova, superior ao reino. Assim, eles passam a dividir a vida em 3 domínios: primeiramente dividindo o antigo reino Monera em dois domínios, o das (i) bactérias e o das recém-descobertas (ii) arqueobactérias, agora já rebatizadas simplesmente como arqueas. O terceiro domínio foi considerado como o dos organismos (iii) eucariotos (que englobavam todos os outros antigos reinos: protozoários, fungos, animais e plantas). Essa hierarquia de domínios tem sido usada com sucesso desde então. Aplicando o princípio darwiniano a essa ideia, sugeriu-se que esses três grandes domínios da vida deveriam ter tido um ancestral comum no passado (Figura 3). A esse ancestral foi dado o nome de LUCA e ele teria dado origem aos três domínios: as arqueobactérias, as eubactérias e os eucariotos (Woese, 1998). O ancestral LUCA teria sido possivelmente um organismo que já apresentava constituição celular e que teria emergido a partir da sopa primordial.
Décadas foram passando e os estudos sobre as arqueas avançaram muito. Descobriu-se, por um lado, que elas também podiam ser encontradas em ambientes comuns; além de que se descobriu também que existiam bactérias que viviam em ambientes extremos. Décadas de estudos demonstraram que esses dois grupos de organismos procarióticos (desprovidos de membrana nuclear) teriam características bem distintas, tanto no genoma, quanto na membrana e em sua bioquímica.
Nas décadas de 1990 e 2000, a biologia avançou rapidamente para a elucidação das sequências completas dos genomas (Davies, 2001). Isso permitiu um estudo comparativo entre os genomas completos de inúmeras bactérias, arqueas e eucariotos. Esses estudos demonstraram que os eucariotos poderiam ser de fato considerados como arqueas mais sofisticadas que haviam realizado possivelmente múltiplas eventos de simbiose com bactérias e vírus, permitindo o surgimento do envelope nuclear e das organelas com membrana, como as mitocôndrias e os cloroplastos. Pesquisadores interessados na origem da vida então almejavam descobrir qual seria o genoma do ancestral comum entre bactérias e arqueas. Diversos estudos foram feitos para identificar qual teria sido a mais provável constituição genômica para o LUCA e esses trabalhos eram feito a partir da observação de quais seriam os genes que estariam presentes em dezenas ou até centenas de organismos oriundos dos 3 domínios (Farias e Prosdocimi, 2017). Assim, ao encontrar os genes presentes tanto nas (i) arqueas, quanto (ii) nas bactérias e (iii) nos eucariotos, os pesquisadores almejavam descobrir qual seria a constituição molecular básica do LUCA. A ideia era que, se um gene estava presente em organismos dos três domínios, logo ele deveria ser oriundo do ancestral comum entre eles, ou seja, o LUCA.
Trabalhos assim começaram a sair em meados da década de 1990, chefiados por pesquisadores que almejavam continuar os trabalhos de Woese, Miller e outros grandes cânones da pesquisa em origem da vida. No ano de 2016, estávamos fazendo uma revisão sobre esses trabalhos para tentar entendê-los melhor, quando uma publicação importante saiu na revista Nature Microbiology. O trabalho era dirigido pelo alemão William Martin e usava métodos bastante confiáveis para demonstrar que existiam pelo menos 355 genes compartilhados entre os 3 domínios da vida (Weiss et al., 2016). Analisando esses genes, Martin e seus colaboradores apresentaram uma proposta de um LUCA celular, anaeróbico e que vivia em ambientes extremos, sendo capaz de produzir energia através de quimiossíntese.
Ficamos muito impressionados com esse trabalho por vários motivos, mas o que mais nos surpreendia era pensar: como um organismo ancestral poderia ter centenas genes? As forças de auto-organização molecular operando em moléculas simples de ácidos nucleicos e proteínas na Terra primitiva não poderiam produzir três centenas de genes de uma hora para outra! O LUCA, portanto, já era um organismo altamente complexo. Ao constatarmos essa obviedade, entendemos que a origem da vida não era um fenômeno que estava ligado a esse tipo de pesquisa. Esse foi o primeiro ponto de partida para nossa investigação: entendemos que os estudos sobre a origem da vida estavam muito distantes desses estudos, que tratavam mais sobre a origem da célula. A célula possivelmente teria se originado quando os genomas apresentavam pouco mais de 300 genes. Mas a origem da vida, para nós, deveria ter acontecido quando ainda os primeiros genes estariam se formando. Definimos então nosso primeiro dogma ou paradigma científico que queríamos tratar: a origem da vida não era a origem da célula (Prosdocimi, Jheeta e Farias, 2018). Embora possa parecer óbvio quando dito assim, os trabalhos científicos publicados até hoje não fazem claramente essa distinção conceitual. E isso gera muita confusão no campo.
O FUCA é o ancestral da vida
Com essa constatação, decidimos então partir para investigar o que seria conhecido sobre a constituição dos primeiros genes, quais seriam eles e como teriam se desenvolvido. Antes de tudo, precisaríamos entender o que seria um gene. O conceito de gene é até hoje alvo de inúmeras controvérsias e de um debate acalorado nos círculos acadêmicos (Keller, 2000). Em uma lenda muito difundida na área, diz-se que um prêmio Nobel disse certa vez “se você pergunta o que é um gene, você nunca vai saber”, sugerindo que a complexidade do gene é tanta que apenas os especialistas que tratam deles é que podem entender e que, mesmo eles, são incapazes de descrevê-los em palavras. De fato, não há hoje um conceito inequívoco e universal sobre o que seja um gene, embora haja sim um consenso entre os especialistas que ele seja um tipo de informação escrita a partir da sequência de nucleotídeos na linguagem química de um ácido nucleico (RNA ou DNA).
Já vimos que, para que exista um gene codificador de proteínas, é necessário existir uma codificação genética. Por isso, ao investigarmos a origem do primeiro gene, devemos entender como aconteceu a emergência do próprio código genético. O código genético é um conjunto de regras e instruções que determina como as informações genéticas contidas em uma molécula de ácido nucleico deverão convertidas em proteínas funcionais. Ou seja, é uma linguagem que traduz a sequência de nucleotídeos de um ácido nucleico, em trincas, para a uma sequência de aminoácidos em um peptídeo. O código genético é universal na maioria dos organismos vivos, o que significa que as mesmas sequências de três nucleotídeos (os códons) codificam os mesmos aminoácidos em diferentes espécies. Como já comentamos, a origem desse código é um dos problemas mais complexos e fascinantes da biologia e sua elucidação pode render incríveis honrarias científicas. Embora o completo entendimento de sua origem e auto-organização ainda nos escape, sabemos de muitas coisas sobre como ele se desenvolveu. A base para o estudo da origem do código genético está ligada ao entendimento sobre a origem e evolução de uma organela ribonucleoproteica conhecida como ribossomo. O ribossomo é uma organela celular altamente complexa formada por vários RNAs e dezenas de proteínas.
Os ribossomos encontrados hoje nos organismos biológicos consistem em máquinas moleculares altamente complexas, normalmente formadas por algumas poucas moléculas de RNA e algumas dezenas de moléculas de proteínas (Yonath, Yusupov e Yusupova, 2000)(Figura 4). Entretanto, o ribossomo possui uma partezinha muito particular que tem uma importância fundamental. Essa parte é o seu centro catalítico e basicamente consiste em um RNA enovelado em uma certa estrutura tridimensional que o permite catalisar a ligação entre dois aminoácidos. Um centro catalítico refere-se a uma região específica de uma molécula, geralmente uma enzima ou ribozima, que facilita a ocorrência de uma reação química específica. É a parte específica da molécula onde as interações eletrostáticas, ligações covalentes ou outras interações químicas acontecem para acelerar a reação química. Nas enzimas, o centro catalítico é muitas vezes uma estrutura tridimensional especializada que facilita a ligação do substrato (a molécula sobre a qual a enzima atua) e orienta as moléculas para que a reação ocorra de maneira mais eficiente. As enzimas são conhecidas por suas capacidades catalíticas específicas, e o centro catalítico é crucial para esse processo. Essas regiões desempenham um papel essencial na aceleração de reações químicas específicas, tornando-as mais favoráveis em condições biológicas. No ribossomo, o centro catalítico é chamado de centro de transferência de peptídeos (do inglês, para Peptidyl-Transferase Center, ou PTC) (Schmeing et al., 2005; Yusupov et al., 2006). Ali, um nucleotídeo de adenina (A) é capaz de catalisar a reação que une dois aminoácidos, desempenhando um papel crucial na formação de ligações peptídicas entre os aminoácidos e resultando na elongação da cadeia polipeptídica, aminoácido por aminoácido.
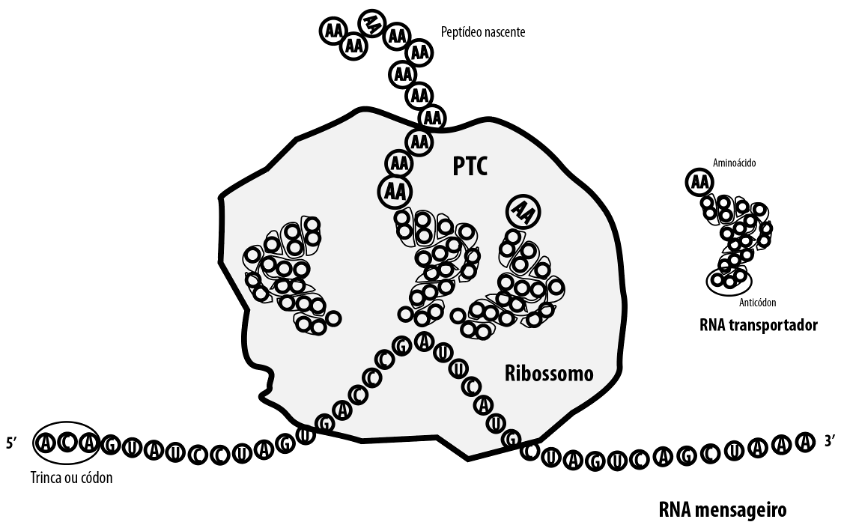
O surgimento desse PTC foi uma etapa fundamental para a origem da vida, pois foi ele que permitiu que um RNA fosse capaz de catalisar a união de aminoácidos para formar peptídeos. Sugere-se que essa união teria sido inicialmente aleatória, ou melhor, quase aleatória porque deveria se basear na abundância relativa de cada tipo de aminoácido presente na sopa primordial (Prosdocimi e Farias, 2023). A partir daí declaramos o nascimento de um proto-organismo fundamental que virá a ser o primeiro organismo vivo logo que todo o sistema de codificação genética estiver montado. Esse proto-organismo nós chamamos de FUCA (do inglês para First Universal Common Ancestor) e esse provável proto-PTC teria sido o primeiro gene a surgir, ou seja, o atrator estranho que coordenou a emergência do fenômeno que hoje chamamos de vida (Prosdocimi, José e Farias, 2019).
Tal mecanismo teria produzido proteínas quase-aleatórias por muito tempo. Aos poucos, entretanto, ele foi se complexificando e o código genético foi sendo montado a partir de uma relação entre diferentes tipos de moléculas de RNAs surgidas nesse contexto. Num primeiro momento, acreditamos que um mesmo RNA, ou seja, o proto-PTC teria atuado como RNA mensageiro, ribossômico e transportador (Prosdocimi e Farias, 2019). De fato, o RNA transportador é o menor, enquanto o RNA mensageiro e o RNA ribossômico teriam sido formados pela concatenação de diversos RNAs transportadores (Prosdocimi et al., 2020).
Conclusões
A origem da vida não é origem das células. Por muito tempo os biólogos tentaram entender a história evolutiva das células, porém já está claro que a origem da vida aconteceu muito antes e esteve relacionada à origem do primeiro gene e de uma interação simbiótica entre ácidos nucleicos e proteínas (Prosdocimi, José e Farias, 2021). Essa teoria marguliana sugere que a simbiose química teria emergido quando um pedaço de RNA autorreplicante (o proto-PTC) foi capaz de ligar aminoácidos consecutivamente através de ligações covalentes, organizando a produção de peptídeos e permitindo a produção de grandes polímeros protéicos. Desde então, esse mecanismo de codificação de proteínas a partir de moldes de ácidos nucleicos se desenvolveu e se complexificou através de um mecanismo de auto-organização ainda pouco conhecido, até formar um código genético primitivo. Foi a organização desse código molecular de intercâmbio de informações entre moléculas de ácidos nucleicos e proteínas que permitiu a emergência dos sistemas biológicos (Prosdocimi e Farias, 2019).
A vida deve ser entendida, dessa forma, como um código químico processual que se auto-organizou na Terra primitiva (provavelmente no período hadeano) e que vem se transformando dinamicamente desde então. Esse código químico, entretanto, não é um código químico qualquer, porém um código tal que foi automontado através da interação mutualística entre dois tipos de macromoléculas em especial: os ácidos nucleicos e os peptídeos (ou proteínas). Portanto, dizemos que a essência mais fundamental da vida se baseia em um processo ribonucleoproteico (Farias e Prosdocimi, 2022). Esse processo deu origem a um proto-organismo que chamamos de FUCA e que foi o primeiro organismo vivo na Terra (Prosdocimi, José e Farias, 2019). A partir daí, o FUCA se reproduziu e deu origem a diferentes de populações de ribonucleoproteínas autorreplicantes que produziam proteínas organizadas e que se interagiam com o meio ambiente.
A partir da seleção natural operando em nível molecular, cada uma dessas populações de descendentes do FUCA foi otimizando sua interação com diferentes moléculas do meio ambiente. Essas populações herdeiras do FUCA foram chamadas de progenotos (de acordo com um conceito definido originalmente por Carl Woese) e diferentes populações de progenotos foram responsáveis pela incorporação de vias químicas já presentes na sopa primitiva dentro dos organismos biológicos (Prosdocimi e Farias, 2022). Essas vias protobióticas permitiram o desenvolvimento dos progenotos e a complexificação de seus genomas e de suas interações com o meio ambiente (Prosdocimi e Farias, 2023). Em algum momento mais avançado, muitos desses progenotos também entraram em um processo de simbiose, passando a viver juntos e colaborar, formando assim o ancestral de todas as células, que foi o LUCA. Nossa teoria sugere que o LUCA ainda era um progenoto “desnudo”, ou seja, um organismo sem membrana plasmática formada por lipídeos (Farias, José e Prosdocimi, 2021). Tanto as vias que produziram a membrana quanto aquelas que produziram o DNA parecem ter sido as últimas a serem incorporadas em diferentes populações do LUCA, dando assim origem às bactérias e às arqueas (Prosdocimi e Farias, 2023).
Tendo emergido a partir da auto-organização de um código molecular que permitiu a simbiose entre dois tipos de macromoléculas abundantes na sopa primordial, a vida se desenvolveu através da aquisição de outros códigos que se sobrepuseram a esse primeiro. Por isso, dizemos que a vida é baseada em um macrocódigo de múltiplas camadas (Farias, Prosdocimi e Caponi, 2021; Prosdocimi e Farias, 2021).
Enquanto este código de múltiplas camadas representa a vida tal qual conhecemos, não podemos saber se existirão outros tipos de fenômenos vivos que auto-organizarão códigos diferentes. Porém, parece-nos que a formação de códigos contingentes auto-organizados é o principal fenômeno que identifica a vida na Terra. Apenas se encontrarmos outros tipos de organismos vivos é que poderemos teorizar sobre suas organizações moleculares. Na Terra, tanto os vírus quanto as células possuem esses macrocódigos de múltiplas camadas e são capazes de imprimir proteínas usando um código escrito na linguagem dos ácidos nucleicos. Embora esse chamado código genético ribonucleoproteico tenha dado o ponta-pé original para a emergência do fenômeno vida, hoje a vida é composta por códigos bastante mais complexos que se sobrepuseram a este primeiro código. Este conceito é necessário e suficiente para definir todo o fenômeno vida que observamos na Terra e, portanto, nos parece o mais adequado a ser utilizado.
Epílogo
Este trabalho começou como uma amizade dos tempos de doutorado, quando ainda não tínhamos completado três décadas de existência. Meu amigo Sávio Torres de Farias viria a ser o primeiro doutor em Genética pela UFMG e eu, Francisco Prosdócimi, o primeiro doutor em bioinformática pela mesma instituição. Defendemos nossos doutorados em 2006, ambos com 27 anos. À época, Sávio já trabalhava com a origem do código genético, enquanto Francisco se dedicava em estudar os genomas de organismos com importância em biomedicina e biodiversidade.
Em 2014, Sávio organizou um evento internacional de biologia evolutiva em João Pessoa e convidou pesquisadores altamente renomados, como o físico teórico estadunidense Stuart Kaufmann (1939-) e a pesquisadora israelense laureada com prêmio Nobel, Ada Yonath (1939-). Eu também fui convidado e, assim, começamos nossa parceria ao desenvolvermos modelos teóricos (baseados em observações e dados empíricos) para o campo da origem da vida. Essa amizade e parceria frutífera já rendeu dezenas de trabalhos científicos, viagens, pós-doutorados no Brasil e no México, além de apresentações em congressos por todo o mundo.
Agradecimentos
Gostaria de agradecer à FAPERJ pela bolsa de cientista do nosso estado (CNE E-26/200.940/2022), ao CNPq pela bolsa de pesquisador (306346/2022-2). Agradeço ao amigo Nelson Job, que inspirou a escrita do presente artigo e o avaliou em seus estágios iniciais e ao parceiro de tantos anos e trabalhos científicos, Sávio Torres de Farias, que também leu o presente ensaio e sugeriu pequenas mudanças.
Referências
Barbieri, M. From Biosemiotics to Code Biology. Biol Theory 9, 239–249 (2014a). https://doi.org/10.1007/s13752-013-0155-6.
Bergh O, Børsheim KY, Bratbak G, Heldal M. High abundance of viruses found in aquatic environments. Nature. 1989 Aug 10;340(6233):467-8.
Comeau AM, Hatfull GF, Krisch HM, Lindell D, Mann NH, Prangishvili D. Exploring the prokaryotic virosphere. Res Microbiol. 2008 Jun;159(5):306-13. doi: 10.1016/j.resmic.2008.05.001. Epub 2008 May 29. PMID: 18639443.
Davies, K. (2001). Desvendando o Genoma. São Paulo: Companhia das Letras.
Dupré J (2012) The constituents of life 2: organisms and systems. Processes of life essays in the philoso- phy of biology. Oxford University Press, New York, pp 85–100.
Dupré J, Nicholson D (2018) A manisfesto for a processual pilosophy of biology. Everything flows: towards a processual philosophy of biology. Oxford University Press, Oxford, pp 3–45.
Emmeche C (1998) Defining life as a semiotic phenomenon. Cybern Hum Knowing 5(1):3–17
Enard D, Cai L, Gwennap C, Petrov DA. Viruses are a dominant driver of protein adaptation in mammals. Elife. 2016 May 17;5:e12469. doi: 10.7554/eLife.12469. PMID: 27187613; PMCID: PMC4869911.
Farias ST, Jheeta S, Prosdocimi F (2019) Viruses as a survival strategy in the armory of life. Hist Philos Life Sci 41:45. https://doi.org/10.1007/s40656-019-0287-5
Farias ST, Jose MV, Prosdocimi F. Is it possible that cells have had more than one origin? Biosystems. 2021 Apr;202:104371. doi: 10.1016/j.biosystems.2021.104371. Epub 2021 Jan 30. PMID: 33524470.
Farias ST, Prosdocimi F. Buds of the tree: the highway to the last universal common ancestor. International Journal of Astrobiology. 2017;16(2):105-113. doi:10.1017/S147355041600029X.
Farias ST, Prosdocimi F. RNP-world: The ultimate essence of life is a ribonucleoprotein process. Genet Mol Biol. 2022 Sep 23;45(3 Suppl 1):e20220127. doi: 10.1590/1678-4685-GMB-2022-0127. PMID: 36190700; PMCID: PMC9528728.
Farias ST, Prosdocimi F, Caponi G. Organic Codes: A Unifying Concept for Life. Acta Biotheor. 2021 Dec;69(4):769-782. doi: 10.1007/s10441-021-09422-2. Epub 2021 Jul 30. PMID: 34331153.
Goodwin JT, Mehta AK and Lynn D. (2012). Digital and analog chemical evolution. Acc Chem Res 45(12): 2189-2199.
Keller, EF (2000). O Século do Gene. Rio de Janeiro: Relume Dumará.
Koskella B, Hernandez CA, Wheatley RM. Understanding the Impacts of Bacteriophage Viruses: From Laboratory Evolution to Natural Ecosystems. Annu Rev Virol. 2022 Sep 29;9(1):57-78. doi: 10.1146/annurev-virology-091919-075914. Epub 2022 May 18. PMID: 35584889.
Lanier KA, Petrov AS, Williams LD. The Central Symbiosis of Molecular Biology: Molecules in Mutualism. J Mol Evol. 2017 Aug;85(1-2):8-13. doi: 10.1007/s00239-017-9804-x. Epub 2017 Aug 7. PMID: 28785970; PMCID: PMC5579163.
Oparin, AI (1924). Происхождение жизни (The Origin of Life).
Prosdocimi F, Cortines JR, José MV, Farias ST. Decoding viruses: An alternative perspective on their history, origins and role in nature. Biosystems. 2023 Sep;231:104960. doi: 10.1016/j.biosystems.2023.104960. Epub 2023 Jul 16. PMID: 37437771.
Prosdocimi F, Farias ST. A emergência dos sistemas biológicos: uma visão molecular sobre a origem da vida. ISBN: 978-65-900624-1-3. Editora Artecom Ciência. 2019. 1 ed.
Prosdocimi F, Farias ST. Vírus: reinterpretando a história natural e sua importância. Helius. 2020 v. 3, n. 2, fasc. 3, pp 1791-1811. (https://helius.uvanet.br/index.php/ helius/article/view/186).
Prosdocimi F, Farias ST. Life and living beings under the perspective of organic macrocodes. Biosystems. 2021 Aug;206:104445. doi: 10.1016/j.biosystems.2021.104445. Epub 2021 May 24. PMID: 34033908.
Prosdocimi F, de Farias ST. Entering the labyrinth: A hypothesis about the emergence of metabolism from protobiotic routes. Biosystems. 2022 Oct;220:104751. doi: 10.1016/j.biosystems.2022.104751. Epub 2022 Aug 5. PMID: 35940497.
Prosdocimi F, Farias ST. Origin of life: Drawing the big picture. Prog Biophys Mol Biol. 2023 Apr 18;180-181:28-36. doi: 10.1016/j.pbiomolbio.2023.04.005. Epub ahead of print. PMID: 37080436.
Prosdocimi F, Jheeta S, Torres de Farias S. Conceptual challenges for the emergence of the biological system: Cell theory and self-replication. Med Hypotheses. 2018 Oct;119:79-83. doi: 10.1016/j.mehy.2018.07.029. Epub 2018 Jul 30. PMID: 30122496.
Prosdocimi F, José MV, Farias ST (2019). The First Universal Common Ancestor (FUCA) as the Earliest Ancestor of LUCA’s (Last UCA) Lineage. Chapter 3. In: Pontarotti, P. (eds) Evolution, Origin of Life, Concepts and Methods. Springer, Cham. https://doi.org/10.1007/978-3-030-30363-1_3
Prosdocimi F, José MV, de Farias ST. The Theory of Chemical Symbiosis: A Margulian View for the Emergence of Biological Systems (Origin of Life). Acta Biotheor. 2021 Mar;69(1):67-78. doi: 10.1007/s10441-020-09388-7. Epub 2020 Aug 11. PMID: 32783083.
Prosdocimi F, Zamudio GS, Palacios-Pérez M, Torres de Farias S, V José M. The Ancient History of Peptidyl Transferase Center Formation as Told by Conservation and Information Analyses. Life (Basel). 2020 Aug 5;10(8):134. doi: 10.3390/life10080134. PMID: 32764248; PMCID: PMC7459865.
Roux S, Hallam SJ, Woyke T, Sullivan MB. Viral dark matter and virus-host interactions resolved from publicly available microbial genomes. Elife. 2015 Jul 22;4:e08490. doi: 10.7554/eLife.08490. PMID: 26200428; PMCID: PMC4533152.
Schimmel P, Giege R, Moras D & Yokoyama S. (1993). An operational RNA code for amino acids and possible relationship to genetic code. Proc Nac Ac Sci, 90(19), 8763-8768.
Schmeing TM, Huang KS, Kitchen DE, Strobel SA, Steitz TA. Structural insights into the roles of water and the 2′ hydroxyl of the P site tRNA in the peptidyl transferase reaction. Mol Cell. 2005 Nov 11;20(3):437-48. doi: 10.1016/j.molcel.2005.09.006. PMID: 16285925.
Schrödinger, E (1944). O que é a Vida? O Aspecto Físico da Célula Viva com Mente e Matéria. Fundação Calouste Gulbenkian.
Simons P (2018) Process and precipitates. In: Nicholson DJ, Dupré J (eds) Everything flows: towards a processual philosophy of biology. Oxford University Press, Oxford, pp 49–60
Suttle CA. Viruses: unlocking the greatest biodiversity on Earth. Genome. 2013. 56(10): 542-544. https://doi.org/10.1139/gen-2013-0152.
Yonath A, Yusupov G & Yusupova G. (2000). Crystal structure of the ribosome at 5.5 Å resolution. Science, 289(5481), 905-920.
Yusupova G, Jenner L, Rees B, Moras D, Yusupov M. Structural basis for messenger RNA movement on the ribosome. Nature. 2006 Nov 16;444(7117):391-4. doi: 10.1038/nature05281. Epub 2006 Oct 18. PMID: 17051149.
Wächtershäuser G (1998) Origin of life in an iron – sulfur world. In: Brack A (ed) The molecular origins of life, Cambridge University Press, Cambridge, pp. 206-218.
Watson, JD (1968). The Double Helix: A Personal Account of the Discovery of the Structure of DNA. Atheneum.
Watson, JD & Crick, FHC. (1953). A structure for deoxyribose nucleic acid. In Nature, 171(4356), 737-738.
Weiss MC, Sousa FL, Mrnjavac N, Neukirchen S, Roettger M, Nelson-Sathi S, Martin WF. The physiology and habitat of the last universal common ancestor. Nat Microbiol. 2016 Jul 25;1(9):16116. doi: 10.1038/nmicrobiol.2016.116. PMID: 27562259.
Woese C. The universal ancestor. Proc Natl Acad Sci U S A. 1998 Jun 9;95(12):6854-9.
Woese CR, Fox GE. The concept of cellular evolution. J Mol Evol. 1977a Sep 20;10(1):1-6. doi: 10.1007/BF01796132. PMID: 903983.
Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. Proc Natl Acad Sci U S A. 1977b Nov;74(11):5088-90. doi: 10.1073/pnas.74.11.5088. PMID: 270744; PMCID: PMC432104.